STANDARD
02 32 88 89 90
JOINDRE UN PATIENT
02 32 88 11 11
Plus d’une centaine de personnes ont assisté le 14 mars dernier au second Symposium Post Génomique.
Elles étaient réunies autour d’un même objectif, l’amélioration du diagnostic génomique dans les cancers, les maladies rares, les maladies neurologiques et du développement, grâce à l’utilisation de nouvelles techniques, stratégies et workflows post-génomiques. Les présentations se sont ainsi axées autour du long read, de l’ARN sous toutes ses formes, des études fonctionnelles cellulaires, des organoïdes, de la bioinformatique et des études épigénétiques.Un grand merci à tous les orateurs et tout particulièrement aux conférenciers invités pour la qualité de leurs présentations et les échanges.
Les problématiques de production de données ont aujourd’hui cédé la place à des questionnements aigus sur nos capacités d’interprétation.
En avril 2023, la première édition du Symposium Post Génomique a donc été consacrée aux analyses complémentaires expertes et tests fonctionnels innovants « post-génomiques » dont le but est de caractériser l’impact d’un variant génomique sur la fonction du gène et in fine son implication ou non dans le phénotype.
Une centaine de personnes a assisté à cet évènement, rythmé par des conférences et des sessions de communications orales sélectionnées autour de cette thématique, et clôturé par une table ronde dédiée à la prise en charge et la valorisation des tests fonctionnels.
Congrès organisé par la FHU-G4 Génomique.
9h30 – 10h00 : Accueil café
10h00 – 10h30 : Conférence | Comprehensive haplotype-resolved view of genomic variation and methylation with long-read nanopore sequencing (J. Monlong, Institut de recherche en santé digestive, Toulouse)
10h30 – 11h30 : Communications orales sélectionnées session 1 (long read)
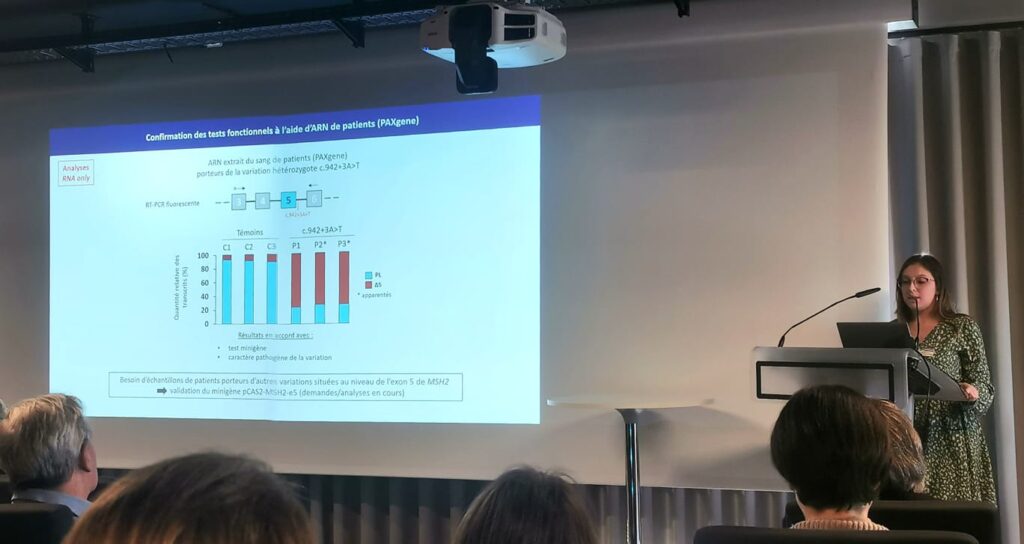
11h30 – 12h00 : Conférence | Tests fonctionnels à haut débit, principes et application au cas du domaine exonucléase du gène POLE (N. Hamzaoui, Institut Cochin, Paris)
12h00 – 12h45 : Communications orales sélectionnées session 2, première partie (tests fonctionnels)
12h45 – 14h00 : Cocktail déjeunatoire
14h00 – 14h30 : Conférence | Organoïdes rétiniens dérivés de cellules iPS humaines : des modèles 3D pour une meilleure compréhension des maladies et le développement de thérapies innovantes – Olivier Goureau, Institut de la vision, Sorbonne Université, Paris
14h30 – 15h45 : Communications orales sélectionnées session 2, seconde partie (tests fonctionnels)
15h45 – 16h15 : Pause
16h15 – 16h45 : Communications orales sélectionnées session 3 (bioinformatique)
16h45 – 17h15 : Communications orales sélectionnées session 4 (épigénétique)
17h30 : Clôture
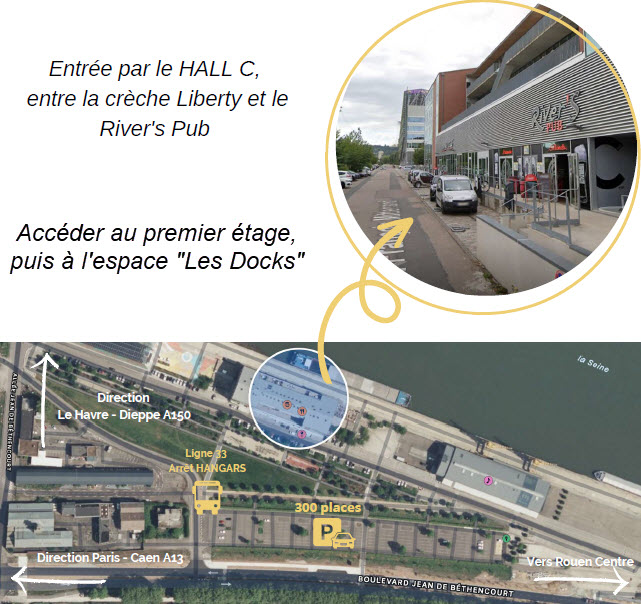
Le congrès se tiendra au Village by CA Rouen Vallée de Seine, implanté au Hangar 107 sur les quais de Seine.
ADRESSE
107 Allée François Mitterrand 76100 Rouen
ACCES DEPUIS LA GARE (2,5 kms)
ACCES EN VOITURE :
Proximité immédiate de l’A13 (Sud III) et de l’A150 (Pont Flaubert).
Stationnement : 350 places gratuites.
EN VELO :
Station Cy’clic et abris.
Ouvert du 01/12/24 au 03/02/25.
Communications orales (15 minutes) uniquement.

Hôpital Charles-Nicolle
37 boulevard Gambetta – 76031 Rouen
Hôpital de Bois Guillaume
147 avenue du Maréchal Juin – 76230 Bois-Guillaume
Hôpital Saint Julien
rue Guillaume Lecointe – 76140 Le Petit-Quevilly
Hôpital de Oissel
rue Pierre Curie – 76350 Oissel
Hôpital de Boucicaut
13 rue Boucicaut – 76130 Mont-St-Aignan
Services d'urgences
116 117
32 37
02 35 15 81 27
144
SAMU